Wissenschaftler schockiert über die erste Beobachtung, dass ein Virus an einem anderen haftet – „Ich kann das nicht glauben“
Die Forscher entdeckten eine einzigartige virale Interaktion, bei der Satellitenphagen physisch an den Helferphagen binden. Diese bahnbrechende Entdeckung, die aus einer zunächst angenommenen seriellen Kontamination resultierte, enthüllt eine neue Art viraler Beziehung und weist auf eine langfristige Koevolution zwischen den beiden Viren hin.
Die Ergebnisse deuten darauf hin, dass viele ähnliche Systeme entdeckt werden könnten.
Niemand hat jemals einen gesehen Virus Ich klammerte mich an einen anderen Virus, bis anomale Sequenzierungsergebnisse das UMBC-Team in die Irre führten und zu einer einzigartigen Entdeckung führten.
Viren und ihre komplexen Zusammenhänge
Es ist bekannt, dass einige Viren, sogenannte Satelliten, nicht nur auf ihren Wirtsorganismus angewiesen sind, um ihren Lebenszyklus abzuschließen, sondern auch auf ein anderes Virus, das als „Helfer“ bekannt ist, erklärt Evan Erel, Professor für Biowissenschaften. Das abhängige Virus benötigt den Helfer entweder zum Aufbau seines Kapsids, einer Schutzhülle, die das genetische Material des Virus umgibt, oder um ihm bei der Replikation zu helfen. DNA.
Diese viralen Beziehungen erfordern, dass sich Satellit und Helfer zumindest vorübergehend in unmittelbarer Nähe zueinander befinden. Bisher sind jedoch keine Fälle bekannt, in denen ein Satellit tatsächlich am Helfer haften geblieben wäre.

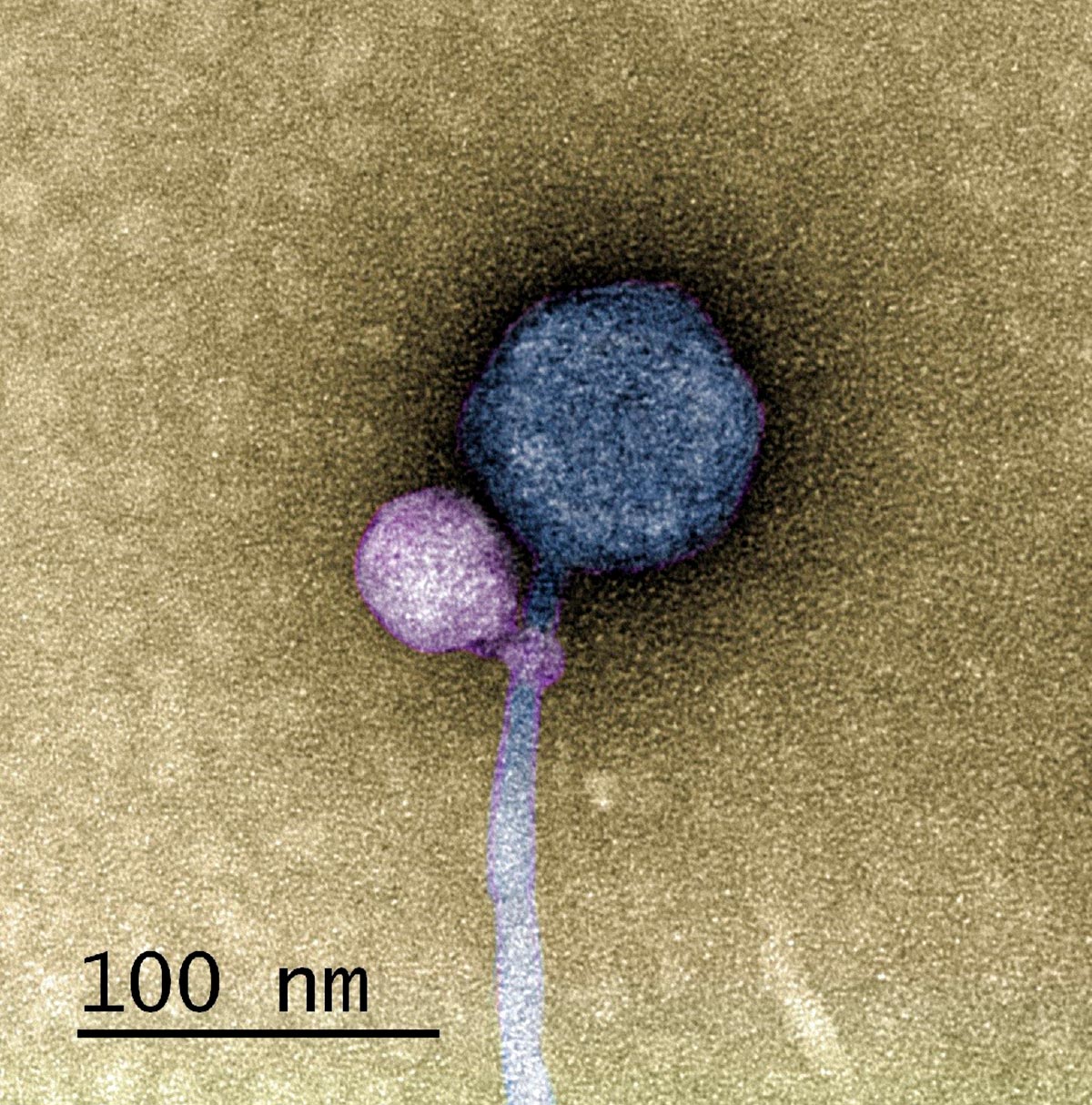
Dieses Farbtransmissionselektronenmikroskopbild zeigt einen neu entdeckten Satellitenvirus, der an den Helfervirus gebunden ist. Diese Forschung stellt das erste Mal dar, dass Wissenschaftler einen Zusammenhang zwischen einem Virus und einem anderen beobachten konnten. Von den 50 beobachteten Assistenten waren 40 per Satellit verbunden. Die bioinformatische Analyse der Genome von Satelliten- und Helferviren liefert Hinweise darauf, warum sich der Satellit so entwickelt hat, dass er mit dem Helfer assoziiert ist, und legt nahe, dass sich dieses Paar möglicherweise vor etwa 100 Millionen Jahren gemeinsam entwickelt hat. Bildnachweis: Tajide de Carvalho
Wegweisende Beobachtung
In einem Artikel veröffentlicht in Zeitschrift der International Society of Microbial EcologyDas UMBC-Team und Kollegen von der Washington University in St. Louis (WashU) beschreiben die erste Beobachtung eines Satellitenbakteriums (ein Virus, das Bakterienzellen infiziert), das sich an seinem „Hals“ – dort, wo das Kapsid mit dem Schwanz verbunden ist – dauerhaft an Helferbakterien bindet vom Virus.
In detaillierten elektronenmikroskopischen Bildern, die von Tajid de Carvalho, stellvertretender Direktor der Kerneinrichtungen des College of Natural and Mathematical Sciences und Erstautor der neuen Arbeit, aufgenommen wurden, waren 80 Prozent (40 von 50) der Helfer an einem Satelliten im Nacken befestigt . Einige von denen, die keine zusätzlichen Ranken im Nacken hatten. Eryl, einer der leitenden Autoren der Studie, beschreibt sie als „Bissspuren“.

Tajide de Carvalho in der Keith R. Porter Photography Facility. deCarvalho nutzte die Transmissionselektronenmikroskopie (TEM) der Einrichtung, um atemberaubende Bilder des satellitengestützten Virussystems aufzunehmen, das in der neuen Arbeit besprochen wird. Bildnachweis: Marlayna Dimond ’11/UMBC
„Als ich das sah, sagte ich: ‚Das kann ich nicht glauben‘“, sagt De Carvalho. „Niemand hat jemals gesehen, dass sich ein Phagen – oder ein anderes Virus – an ein anderes Virus anheftet.“
Langfristige virale Partnerschaft
Nach ersten Beobachtungen analysierte Ilya Mascolo, ein Doktorand in Eryls Forschungsgruppe und Co-Erstautor der Arbeit, das Satelliten-, Helfer- und Wirtsgenom und enthüllte weitere Beweise für diese noch nie dagewesene virale Beziehung. Die meisten fremden Viren enthalten ein Gen, das es ihnen ermöglicht, sich nach dem Eindringen in die Zelle in das genetische Material der Wirtszelle zu integrieren. Dadurch kann sich der Satellit reproduzieren, wenn von da an zufällig ein Helfer die Zelle betritt. Die Wirtszelle kopiert bei der Teilung auch die Satelliten-DNA zusammen mit ihrer eigenen DNA.

Evan Erel ist Co-Leiter des SEA-PHAGES-Programms an der UMBC. Der Schwerpunkt seiner Forschungsgruppe liegt auf der Bioinformatik. Bildnachweis: Marlayna Dimond ’11/UMBC
Die WashU-Bakterienprobe enthält außerdem einen Helfer und einen Satelliten. Der Washoe-Satellit verfügt über ein Integrase-Gen und ist nicht direkt mit seinem Helfer verwandt, ähnlich wie zuvor beobachtete Satelliten-Helfersysteme.
Allerdings ist der Satellit in der UMBC-Probe, den die isolierten Studenten MiniFlayer nannten, der erste bekannte Fall eines Satelliten ohne Integrationsgen. Da es sich nicht in die DNA der Wirtszelle integrieren kann, muss es sich jedes Mal, wenn es in die Wirtszelle eindringt, in der Nähe seines Helfers – dem MindFlayer – aufhalten, um zu überleben. Angesichts dessen und obwohl das Team diese Erklärung nicht direkt bewiesen hat, „macht die Bindung jetzt vollkommen Sinn“, sagt Irel. „Wie wollen Sie sonst sicherstellen, dass Sie beide gleichzeitig in die Zelle gelangen?“ „
Zusätzliche bioinformatische Analysen wurden von Mascolo und Julia Lopez Pérez, einer weiteren Doktorandin, durchgeführt. Einer der Studenten, die mit Eryl arbeiteten, verriet, dass MindFlayer und MiniFlayer schon seit langem gemeinsam entwickeln. „Dieser Satellit hat sein Genom so abgestimmt und optimiert, dass es mindestens 100 Millionen Jahre lang mit dem Helfer assoziiert ist“, sagt Erel und deutet an, dass es möglicherweise noch viele weitere Beispiele dieser Art von Beziehung gibt, die darauf warten, entdeckt zu werden. .
Versehentliche Kontamination oder wichtige Entdeckung?
Diese bahnbrechende Entdeckung hätte leicht übersehen werden können. Das Projekt begann als Pilotkurs im SEA-PHAGES-Programm, einem Forschungslehrplan, bei dem Studenten im Grundstudium Phagen aus Umweltproben isolieren, sie zur Sequenzierung schicken und dann Bioinformatik-Tools verwenden, um die Ergebnisse zu analysieren. Als das Sequenzierungslabor der University of Pittsburgh eine Kontamination in einer Probe der University of British Columbia meldete, die den MindFlayer-Phagen enthalten sollte, begann die Reise.

Stephen Caruso ist Co-Leiter des SEA-PHAGES-Programms an der UMBC. Er führt auch Bildungsforschung durch, um den naturwissenschaftlichen Unterricht zu verbessern. Bildnachweis: Marlayna Dimond ’11/UMBC
Die Probe enthielt eine große Sequenz: den erwarteten Phagen. „Aber anstatt das zu entdecken, haben wir auch eine kleine Sequenz gefunden, die mit nichts übereinstimmt, was wir wissen“, sagt Erel, der auch einer der Leiter der Forschung ist. Das SEA-PHAGES-Programm der UMBC mit dem Namen Phage HuntersMit Stephen Caruso, Dozent für Biowissenschaften. Caruso 94, Ph.D. ’02, Biological Sciences, führte die Isolierung erneut durch, schickte sie zur Sequenzierung – und erhielt ähnliche Ergebnisse.
Zu diesem Zeitpunkt zog das Team deCarvalho hinzu, um sich ein Bild davon zu machen, was mit einem Transmissionselektronenmikroskop (TEM) in der Keith R. Porter Imaging Facility (KPIF) der UMBC vor sich geht. Ohne die Bilder wäre eine Entdeckung unmöglich gewesen.
„Nicht jeder verfügt über ein TEM-Gerät“, betont de Carvalho. Aber mithilfe der Tools am KPIF, sagt de Carvalho, „kann ich einige dieser Beobachtungen weiterverfolgen und sie mithilfe von Bildgebung validieren. Es gibt Entdeckungselemente, die wir nur mit TEM erreichen können.“
Implikationen und zukünftige Forschung
Die Entdeckung des Teams ebnet den Weg für zukünftige Arbeiten, um herauszufinden, wie der Satellit verbunden ist, wie häufig das Phänomen auftritt und mehr. „Es ist möglich, dass viele der Phagen, die man für Kontaminanten hielt, tatsächlich Satelliten-Helfersysteme waren“, sagt De Carvalho. „Mit diesem Papier können die Menschen nun möglicherweise mehr über diese Systeme erfahren.“
Referenz: „Gleichzeitiger Eintritt als Anpassung an die Virulenz in einem neuen Streptomyces-infizierenden Satelliten-Helfersystem.“ Klassifizieren„Von Tajidi De Carvalho, Elia Mascolo, Steven M. Caruso, Julia Lopez Perez, Kathleen Weston Haver, Christopher Shaffer und Evan Erel, 31. Oktober 2023, ISME-Magazin.
doi: 10.1038/s41396-023-01548-0

„Böser Kaffee-Nerd. Analyst. Unheilbarer Speckpraktiker. Totaler Twitter-Fan. Typischer Essensliebhaber.“
