Wissenschaftler haben eine überlegene natürliche Immunität gegen SARS-CoV-2 gegen 23 verschiedene Arten identifiziert
Ein Team internationaler Wissenschaftler hat kürzlich hochpotente Anti-Coronavirus 2 (SARS-CoV-2) Antikörper von rekonvaleszenten Spendern identifiziert.
Antikörper sind in der Lage, selbst bei Konzentrationen im Sub-Nanometer-Bereich eine Vielzahl von SARS-CoV-2-Varianten zu neutralisieren. Darüber hinaus verringern Kombinationen dieser Antikörper das Risiko, unkontrollierte Mutationen zu bilden im Labor. Die Studie wurde in der Zeitschrift veröffentlicht Wissenschaft.
Hintergrund
Das schwere akute respiratorische Syndrom Coronavirus 2 (SARS-CoV-2), der Erreger, der die Coronavirus-Krankheit 2019 (COVID-19) verursacht, ist ein umhülltes, positiv-sinniges, einzelsträngiges RNA-Virus, das zur Familie der humanen Beta-Coronaviren gehört. Das erhöhte Glykoprotein auf der Virushülle besteht aus zwei Untereinheiten, S1 und S2. Von diesen bindet die S1-Untereinheit direkt an den Angiotensin-Converting-Enzym-2-(ACE2)-Rezeptor der Wirtszelle über die Rezeptorbindungsdomäne (RBD), um den viralen Eintrittsprozess einzuleiten.
Die meisten therapeutischen Antikörper gegen SARS-CoV-2 werden basierend auf der ursprünglichen Spike-Proteinsequenz entwickelt, die im ursprünglichen Wuhan-Stamm von SARS-CoV-2 gefunden wurde. Somit würden neue virale Varianten mit mehreren Proteinmutationen wahrscheinlich eine Resistenz gegen diese Antikörper entwickeln. In diesem Zusammenhang haben Studien gezeigt, dass Antikörper, die als Reaktion auf derzeit verfügbare COVID-19-Impfstoffe entwickelt wurden, eine geringere Wirksamkeit bei der Neutralisierung neuartiger besorgniserregender Varianten (VOCs) für SARS-CoV aufweisen, einschließlich B.1.1.7, B.1.351 und P1 und b have 1.617.2.
In der aktuellen Studie isolierten und charakterisierten Wissenschaftler Anti-Elevations-RBD-Antikörper von genesenen COVID-19-Patienten.
Identifizierung des Antikörpers
Antikörper wurden von vier rekonvaleszenten Spendern isoliert, die mit dem Washington-1 (WA-1) SARS-CoV-2-Stamm infiziert waren. Die Spike-Sequenz im WA-1-Stamm ähnelt der Spike-Sequenz im ursprünglichen Wuhan-Stamm.
B-Zellen, die aus Blutproben von Spendern isoliert wurden, wurden zur Antikörperidentifizierung sortiert. Dies führte zur Identifizierung von vier neutralisierenden potenten Antikörpern, die auf Spike-RBD abzielen. Diese Antikörper zeigten selbst bei nanomolaren Konzentrationen eine hohe Affinität für SARS-CoV-2.
Um zu bestimmen, ob hochwirksame Antikörper die ACE2-Spike-Bindung hemmen könnten, wurden ACE2-Interferenzassays und Zelloberflächenbindungsassays durchgeführt. Die Ergebnisse zeigten, dass von den 4 Antikörpern zwei an RBDs in der „Up-Position“ und zwei an RBDs in der „Lower-Position“ gebunden waren. Darüber hinaus hemmten drei von vier Antikörpern die RBD-ACE2-Interaktion direkt und einer die Reaktion indirekt durch die sterische Hinderung – die Verlangsamung chemischer Reaktionen aufgrund der sterischen Masse.
Neutralisation durch Antikörper
Alle experimentellen Antikörper zeigten eine signifikant höhere Wirksamkeit bei neutralisierenden Varianten, die die D614G-Mutation des WA-1-Stammes enthielten. Eine zusätzliche Analyse pseudotypisierter lentiviraler Partikel mit Spike-Varianten zeigte, dass die Antikörper eine hohe Wirksamkeit bei der Neutralisierung einer Vielzahl von 10 Spike-Varianten beibehalten.
Wichtig ist, dass drei von vier experimentellen Antikörpern eine hohe Wirksamkeit bei der Neutralisierung von 13 Arten von zirkulierenden Varianten von Interesse/von Interesse für SARS-CoV-2 zeigten, einschließlich B.1.1.7, B.1.351, B.1.427 und B. .1.429 und B.1.526 1, S. 2, B.1.617.1, B.1.617.2.
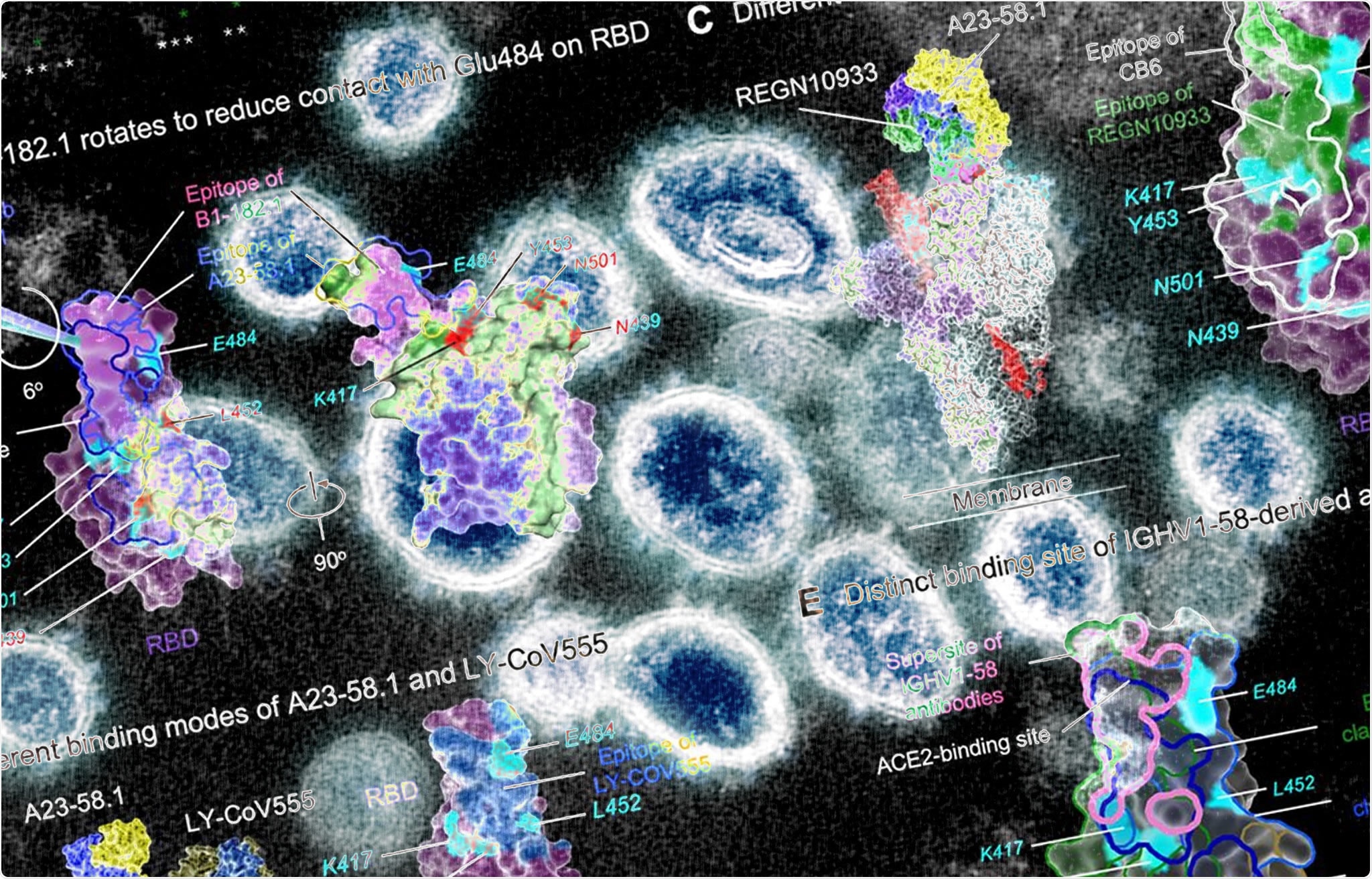
Struktur- und Funktionsanalyse von Antikörpern
Kryoelektronenmikroskopische Analysen der Antikörper- und Antigenkomplexstrukturen zeigten, dass zwei der Antikörper mit der höchsten äquivalenten Stärke an das Spike-Protein binden, wobei sich alle RBDs in einer „höheren Position“ befinden. Weitere Strukturanalysen ergaben, dass die Epitop-Bindungsmuster von Antikörpern für die hohe Neutralisationswirksamkeit gegen SARS-CoV-2-VOCs verantwortlich sind. Die Fähigkeit, Antikörper zu binden und zu neutralisieren, wurde durch drei Mutationsmutanten, einschließlich F486R, N487R und Y489R, negativ beeinflusst.
Antikörperresistenz
Auf den WA-1-Stamm wurde ein Antikörperselektionsdruck ausgeübt, um potentielle Fluchtmutationen zu identifizieren, die während einer Virusinfektion auftreten könnten. Ein positiver Selektionsdruck wurde ausgeübt, indem das Virus mit steigenden Konzentrationen von Antikörpern inkubiert wurde, um eine Antikörperresistenz zu induzieren.
Bei zwei der stärksten Antikörper wurde einer von einer einzelnen F486S-Mutation negativ beeinflusst und der andere wurde von den F486L-, N487D- und Q493R-Mutationen beeinflusst. Die Q493R-Mutation zeigte jedoch eine geringe Wirkung auf die Bindung und Neutralisation. Zusätzliche Analysen ergaben, dass diese Escape-Mutanten hauptsächlich in zirkulierenden viralen Varianten fehlen, was auf einen fehlenden Selektionsdruck hinweist.
Durch die Durchführung mehrerer Selektionsrunden unter Verwendung von Kombinationstherapien mit zwei Antikörpern wurde beobachtet, dass Antikörperkombinationen das Risiko des Erwerbs einer Runaway-Mutation und der nachfolgenden Entwicklung resistenter Virusvarianten verringern können.

„Böser Kaffee-Nerd. Analyst. Unheilbarer Speckpraktiker. Totaler Twitter-Fan. Typischer Essensliebhaber.“
